Funkcje, typy i przykłady enzymów ograniczenia
- 4512
- 1445
- Pani Waleria Marek
Enzymy restrykcyjne Są to endonukleazy stosowane przez niektóre łuki i bakterie do hamowania lub „ograniczenia” propagacji wirusów w środku. Są one szczególnie powszechne w bakteriach i są częścią ich zagranicznego systemu obrony DNA znanego jako system ograniczenia/modyfikacji.
Enzymy te katalizują podwójny pasek DNA w określonych miejscach, powtarzalny i bez użycia dodatkowej energii. Większość wymaga obecności kofaktorów, takich jak magnez lub inne kationów zróżnicowane, chociaż niektóre wymagają również ATP lub S-adenozyl.
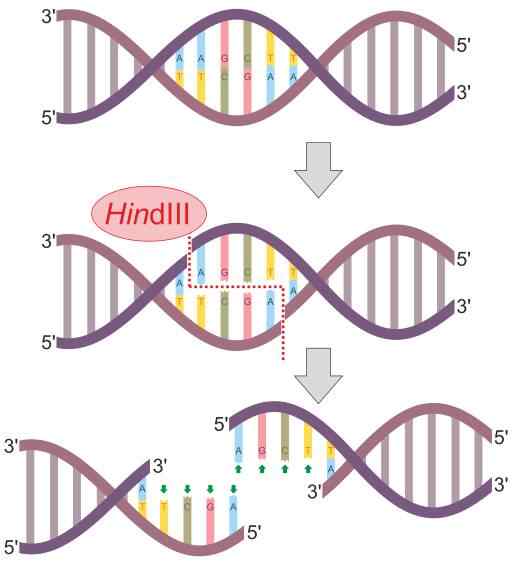
 Hindii Schemat reakcji enzymu enzymu (źródło: helixitta [CC BY-SA 4.0 (https: // creativeCommons.Org/licencje/nabrzeże/4.0)] przez Wikimedia Commons)
Hindii Schemat reakcji enzymu enzymu (źródło: helixitta [CC BY-SA 4.0 (https: // creativeCommons.Org/licencje/nabrzeże/4.0)] przez Wikimedia Commons) Endonucleases ograniczeń zostały odkryte w 1978 roku przez Daniela Nathansa, Arber Wernera i Hamiltona Smitha, którzy otrzymali Nagrodę Nobla w medycynie za ich odkrycie. Jego imię ogólnie wywodzi się z organizmu, w którym są one po raz pierwszy obserwowane.
Takie enzymy są szeroko stosowane w opracowywaniu metod klonowania DNA i innych strategii biologii molekularnej i inżynierii genetycznej. Jego charakterystyka rozpoznawania określonych sekwencji i zdolności cięcia sekwencji zbliżonych do miejsc rozpoznawania sprawiają, że są one potężne narzędzia w eksperymencie genetycznym.
Fragmenty wygenerowane przez enzymy ograniczenia, które działały na określonej cząsteczce DNA, można użyć do odtworzenia „mapy” pierwotnej cząsteczki za pomocą informacji o miejscach, w których enzym przecinał DNA.
Niektóre enzymy restrykcyjne mogą mieć to samo miejsce rozpoznawania w DNA, ale niekoniecznie przecinają je w ten sam sposób. Zatem istnieją enzymy, które sprawiają, że cięcia pozostawiają romos i enzymy, które przecinają skrajne skrajności, które mają różne zastosowania w biologii molekularnej.
Obecnie dostępne są setki różnych enzymów ograniczników, oferowanych przez różne domy komercyjne; Te enzymy działają jako „dostosowane” nożyczki molekularne do różnych celów.
[TOC]
Funkcje
Enzymy restrykcyjne wypełniają przeciwną funkcję polimeraz, ponieważ są one hydrolizowane lub łamie wiązanie estrowe w wiązaniu fosfodiéster między sąsiednimi nukleotydami w łańcuchu nukleotydowym.
W biologii molekularnej i inżynierii genetycznej są to szeroko stosowane narzędzia do budowy wektorów ekspresji i klonowania, a także do identyfikacji określonych sekwencji. Są również przydatne do budowy rekombinowanych genomów i mają duży potencjał biotechnologiczny.
Ostatnie postępy w terapii genowej powodują, że obecne stosowanie enzymów restrykcyjnych do wprowadzenia genów określonych w wektorach, które są pojazdami do transportu takich genów do żywych komórek i że prawdopodobnie mają one zdolność wstawienia się do genomu komórkowego do wykonywania trwałych zmian.
Mechanizm akcji
Enzymy restrykcyjne mogą katalizować podwójne przecięcie DNA, chociaż niektóre są w stanie rozpoznać proste sekwencje DNA pasma, a nawet RNA. Cięcie występuje po rozpoznaniu sekwencji.
Może ci służyć: cykl glioksylanu: charakterystyka, reakcje, regulacja, funkcjeMechanizm działania polega na hydrolizie powiązania fosfodiéster między grupą fosforanową a deoksyrybozą w szkielecie każdego DNA DNA. Wiele enzymów jest w stanie przeciąć w tym samym miejscu, które rozpoznają, podczas gdy inne przecinają 5 do 9 par zasad przed lub po tym samym.
Zwykle enzymy te wycięte na 5 'koniec grupy fosforanowej, powodując fragmenty DNA z końcem fosforycznym 5' i końcem hydroksylowym 3 '.
Ponieważ białka nie mają bezpośredniego kontaktu z miejscem rozpoznawania DNA, należy je przenieść kolejne czas.
Podczas enzymatycznego cięcia powiązanie fosfodiéster każdego z nici DNA jest umieszczone w jednym z aktywnych miejsc enzymów ograniczników. Kiedy enzym opuszcza miejsce rozpoznawania i cięcia, robi to poprzez nieuzasadnione skojarzenia przejściowe.
Chłopaki
Obecnie znane jest pięć rodzajów enzymów ograniczników. Następnie krótki opis każdego z nich:
Enzymy ograniczników typu I
Te enzymy są dużymi białkami pentamerycznymi z trzema podjednostkami, jednym z ograniczeń, metylacji i drugiego do rozpoznawania sekwencji DNA. Te endonukleazy są wielofunkcyjnymi białkami zdolnymi do katalizowania reakcji ograniczenia i modyfikacji, mają ATPASA, a także aktywność topoizomery DNA.
Enzymy tego typu były pierwszymi odkrytymi endonukleazami, zostały one oczyszczone po raz pierwszy w latach 60. i od tego czasu były badane z dużą głębokością.
Enzymy typu I nie są szeroko stosowane jako narzędzie biotechnologiczne, ponieważ miejsce cięcia może znajdować się w zmiennej odległości do 1.000 par zasad dotyczących miejsca rozpoznawania, co czyni je niewiarygodnymi pod względem eksperymentalnej odtwarzalności.
Enzymy ograniczników typu II
Są to enzymy złożone z homodimerów lub tetramerów, które przecinają DNA na miejsca zdefiniowane między 4 a 8 pz długości. Te miejsca do cięcia są zwykle palindromowe, to znaczy rozpoznają sekwencje odczytujące w ten sam sposób w obu kierunkach.
Wiele enzymów ograniczenia typu II w bakteriach przecięło DNA, gdy rozpoznają swój obcy charakter.
Są to prostsze enzymy restrykcyjne, ponieważ nie wymagają żadnego kofaktora innego niż magnez (mg+) do rozpoznawania i przecięcia sekwencji DNA.
Precyzja enzymów restrykcyjnych typu II w rozpoznawaniu i wycinaniu prostych sekwencji w DNA w precyzyjnych pozycjach czyni je jednym z najbardziej używanych i niezbędnych w większości gałęzi biologii molekularnej.
Może ci służyć: Mutualizm: cechy, typy, przykładyW grupie enzymów ograniczników typu II znajduje się wiele podklas sklasyfikowanych według niektórych właściwości, które są unikalne dla każdego. Klasyfikacja tych enzymów jest przeprowadzana przez dodanie liter alfabetu, od A do Z następującego nazwy enzymu.
Niektóre z najbardziej znanych podklas ich użyteczności to:
Podklasa iia
Są to różne podjednostki dímeros. Rozpoznają sekwencje asymetryczne i są używane jako idealne prekursory do generowania enzymów tnąca.
Podklasa IIB
Składają się z jeszcze jednego dimerów i przecinają DNA po obu stronach sekwencji rozpoznawania. W przedziale pary zasad wycinają obie nici DNA.
Podklasa IIC
Enzymy tego typu są polipeptydami z funkcjami podziału i modyfikacji nici DNA. Te enzymy przecinają obie nity asymetrycznie.
Podklasa iie
Enzymy tej podklasy są najczęściej stosowane w inżynierii genetycznej. Mają miejsce katalityczne i ogólnie wymagają efektora Alestro. Te enzymy muszą oddziaływać z dwiema kopiami ich sekwencji rozpoznawania, aby wykonać wydajne cięcie. W tej podklasie znajdują się enzymy EcoRII i ECORI.
Enzymy ograniczników typu III
Endonukleazy ograniczające typu III składają się tylko z dwóch podjednostek, jeden jest odpowiedzialny za rozpoznawanie i modyfikację DNA, podczas gdy drugi jest odpowiedzialny za cięcie sekwencji.
Te enzymy wymagają dwóch kofaktorów do ich działania: ATP i magnezu. Enzymy ograniczników tego typu mają dwa asymetryczne miejsca rozpoznawania, przemieszczają DNA w sposób zależny.
Enzymy ograniczników typu IV
Enzymy typu IV są łatwe do zidentyfikowania, ponieważ wycinają DNA znakami metylacji, składają się z kilku różnych podjednostek, które są odpowiedzialne za rozpoznawanie i cięcie sekwencji DNA. Te enzymy używają kofaktorów GTP i charakterystycznego magnezu.
Specyficzne miejsca cięcia obejmują łańcuchy nukleotydowe z metylowanymi lub hydroksymetycznymi resztami cytozyny w jednym lub obu nici kwasach nukleinowych.
Enzymy ograniczników typu V
Ta klasyfikacja grupuje enzymy typu cisper-cas, które identyfikują i przecinają specyficzne sekwencje DNA organizmów atakujących. Enzymy CAS używają zsyntetyzowanego przewodnika RNA Strand, aby rozpoznać i atakować organizmy atakujące.
Enzymy sklasyfikowane jako typ V są polipeptydami ustrukturyzowanymi według enzymów typu I, II i II. Mogą wyciąć sekcje DNA prawie każdego organizmu i z dużym zakresem. Jego elastyczność i łatwość zatrudnienia sprawiają, że enzymy są jednym z najczęściej używanych narzędzi w inżynierii genetycznej wraz z enzymami typu II.
Może ci służyć: propionibacteriumPrzykłady
Do wykrywania polimorfizmów DNA zastosowano enzymy restrykcyjne, szczególnie w badaniach genetyki populacji i badaniach ewolucyjnych z wykorzystaniem mitochondrialnego DNA, w celu uzyskania informacji o wskaźnikach podstawienia nukleotydów.
Obecnie wektory stosowane do transformacji bakterii z różnymi celami mają miejsca wielokalowe, w których znajdują się miejsca rozpoznawania dla wielu enzymów ograniczników.
Wśród tych enzymów najpopularniejsze są Ecori, II, III, IV i V, uzyskane i opisane po raz pierwszy I. coli; Hindiiii, od H. grypa i Bamhi B. Amyloliquefaciens.
Bibliografia
- BIKLE, t. DO., & Kruger, D. H. (1993). Biologia ograniczenia DNA. Recenzje mikrobiologiczne, 57(2), 434-450.
- Boyaval, s. 1., Moineau, s., Romero, zm. DO., & Horvath, p. (2007). CRISPR zapewnia nabycie przeciwko wirusom w prokariotach. Nauka, 315(Marzec), 1709-1713.
- GOORDELL, d. (2002). Perspektywa molekularna: endonukleaz ogranicznika. Komórki macierzyste podstawy medycyny przeciwnowotworowej, 20, 190-191.
- Halford, s. I. (2001). Przeskakiwanie, skakanie i zapętlanie przez enzymy ograniczników. Transakcje społeczeństwa biochemicznego, 29, 363-373.
- Jeltsch, a. (2003). Utrzymanie tożsamości gatunku i kontrola specjacji bakterii: nowa funkcja systemów ograniczników/modyfikacji? Gen, 317, 13-16.
- Krebs, J., Goldstein, e., & Kilpatrick, s. (2018). Geny Lewina XII (12 wyd.). Burlington, Massachusetts: Jones & Bartlett Learning.
- Li i., Chleb, s., Zhang i., Ren, m., Feng, m., Peng, n.,... ona, Q. (2015). Wykorzystanie systemów CRISPR-CAS typu I i typu III do edycji genomu. Badanie kwasów nukleinowych, 1-12.
- Loenen, w. DO. M., Dryden, d. T. F., Raleight, e. DO., I Wilson, G. G. (2013). Enzymy ograniczników typu I i ich krewni. Badanie kwasów nukleinowych, 1-25.
- Nathans, zm., & Smith, H. ALBO. (1975). Endonukleaz ograniczenia w analizie i restrukturyzacji cząsteczek DNA. Annu. Obrót silnika. Biochem., 273-293.
- Nei, m., & Tajima, F. (1981). Polimorfizm DNA wykrywalny przez endonukleazę restrykcyjną. Genetyka, 145-163.
- Pingoud, a., Fuxreiter, m., Pingoud, v., & Wende, w. (2005). Nauk o życiu komórkowym i molekularnym endonukleaza Ograniczenia typu II: struktura i mechanizm. CMLS nauk komórkowy i molekularny, 62, 685-707.
- Roberts, r. (2005). Jak enzymy ograniczające stały się robotami biologii molekularnej. PNA, 102(17), 5905-5908.
- Roberts, r. J., & Murray, k. (1976). Endonukleaz ograniczenia. Krytyczne recenzje w biochemii, (Listopad), 123-164.
- Stoddard, ur. L. (2005). Struktura i funkcja endonukleazy homowania. Kwartalne recenzje biofizyki, 1-47.
- Tock, m. R., & Dryden, D. T. F. (2005). Biologia ograniczenia i przeciwdziałania. Obecna opinia w mikrobiologii, 8, 466-472. https: // doi.Org/10.1016/j.Moje b.2005.06.003
- Wilson, g. G., & Murray, n. I. (1991). Systemy ograniczeń i modyfikacji. Annu. Obrót silnika. Genet., 25, 585-627.
- Wu, z., & Mou, k. (2016). Genomowe spostrzeżenia na temat wirulencji i genetyki populacji i genetyki populacji. Infec. Dis. Tłumaczyć. Med., 2(3), 109-19.
- Yuan, r. (1981). Struktura i mechanizm wielofunkcyjnych ograniczeń endonukleaz. Annu. Obrót silnika. Biochem., pięćdziesiąt, 285-315.
- « Generowanie cech 1914, autorzy i prace
- Wzajemnie wykluczające się właściwości i przykłady zdarzeń »

