Mechanizmy replikacji DNA, u prokariotów i eukariotów
- 4571
- 1419
- Eliasz Dubiel
Replikacja DNA (Kwas desoksyrybonukleinowy) Składa się z kopiowania genomu, to znaczy wszystkich informacji genetycznych zawartych w DNA organizmu, w celu uzyskania dwóch identycznych kopii. Genom ma niezbędne informacje do zbudowania pełnego organizmu.
Przed podziałem komórki występuje replikacja DNA. Poprzez mejozę występują gamety do reprodukcji płciowej. Poprzez mitozę zachodzi wymiana komórek (na przykład skóra i krew) oraz rozwój (na przykład tkanki i narządy).
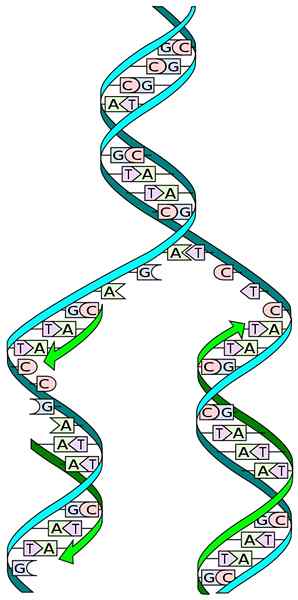
 Źródło: I, MadPrime [CC0]
Źródło: I, MadPrime [CC0] Znajomość struktury DNA pozwala nam zrozumieć sposób, w jaki występuje jej replikacja. Struktura DNA składa się z podwójnego śmigła, złożonego z dwóch łańcuchów antypalalowych kolejnych nukleotydów, których podstawy azotu są uzupełnione konkretnie.
Podczas replikacji każdy z łańcuchów podwójnego łańcucha DNA działa jak pleśń do biosyntezy nowego łańcucha. Dwa nowo zsyntetyzowane łańcuchy mają podstawy uzupełniające się do podstaw łańcucha pleśni: adenina (A) z Timiną (T) i cytozyną (C) z guaniną (G).
Kilka enzymów i białek uczestniczy w replikacji DNA. Na przykład otwieranie podwójnego śmigła DNA, utrzymywanie otwartego DNA i dodanie deoksyrybonukleozydos-5'-trifosforanu (DNTP), aby utworzyć nowy łańcuch.
[TOC]
Replikacja DNA jest półkonserwatywna
W oparciu o strukturę DNA, Watson i Crick zaproponowali, że replikacja DNA zachodzi w sposób pół konserwujący. Zostało to wykazane przez Meselson i Stahl, oznaczając DNA Escherichia coli Z ciężkim izotopem azotu, piętnaścieN, obserwując kilka pokoleń wzór dystrybucji w pożywce hodowlanej z lekkim azotem, 14N.
Messelson i Stahl stwierdzili, że w pierwszej generacji dwie córki miały cząsteczki DNA, każda cząsteczka oznaczona łańcuchem z ciężkim izotopem azotu, a druga z lekkim izotopem. W przeciwieństwie do rodzicielskiej cząsteczki DNA, która miała dwa łańcuchy oznaczone ciężkim izotopem, piętnaścieN.
W drugiej generacji 50% cząsteczek DNA było podobnych do pierwszej generacji, a pozostałe 50% miało tylko azot lekki. Interpretacja tego wyniku polega na tym, że śmigła podwójnej córki ma łańcuch rodzicielski (który działa jako pleśń) i nowy łańcuch.
Półkonserwencyjny mechanizm replikacji implikuje oddzielenie łańcuchów DNA i kojarzenie podstawowych baz za pomocą kolejnych nukleotydów, wytwarzając dwie córki zubełko córek podwójnych córek.
Replikacja w bateriach
Początek replikacji DNA w bakteriach
Bakterie DNA składa się z okrągłego chromosomu i ma tylko jedno miejsce pochodzenia replikacji. Z tego miejsca biosynteza dwóch córek odbywa się dwukierunkowo, tworząc dwa widelce replikacyjne, które poruszają się w kierunku przeciwnym do pochodzenia. Ostatecznie widelce kończą replikację.
Replikacja zaczyna się od połączenia białek DNAA w miejscu pochodzenia. Te białka z kolei tworzą kompleks. Wtedy białka HU i IHF są wiążące.
Następnie łączy się białka DNAC, które sprawiają, że helikazy DNA łączą. Pomagają one zrelaksować DNA i złamać wiązania wodorowe, utworzone w parach zasad. Tak więc dwa łańcuchy dzielą się jeszcze bardziej, tworząc dwa proste łańcuchy.
Może ci służyć: czym jest permutacja chromosomalna?Topoisomeraza II lub DNA Girasa porusza się przed DNA Helicy. Białka wiązania DNA (SSB) z pojedynczym łańcuchem utrzymują oddzielne łańcuchy DNA. Tak więc może rozpocząć się biosynteza łańcucha córki.
Biosynteza łańcuchów DNA córek w bakteriach
Enzym prima jest odpowiedzialny za syntetyzację krótkich łańcuchów RNA zwanych starterami, które mają długość 10 do 15 nukleotydów. Polimeraza DNA zaczyna dodawać 5'-trifosforan (DNTPS) do 3'-OH cukru gruntownego, po czym łańcuch nadal rośnie z tego samego końca.
Ponieważ łańcuchy DNA są antyrównoległe, starter jest syntetyzowany w łańcuchu prowadzenia i wiele starterów w opóźnionym łańcuchu. Z tego powodu biosynteza opóźnionego łańcucha jest nieciągła. Chociaż łańcuchy DNA są antyrównoległe, widelca replikacji porusza się w jednym kierunku.
Polimeraza DNA jest odpowiedzialna za tworzenie wiązań kowalencyjnych między sąsiednimi nukleotydami nowo zsyntetyzowanych łańcuchów, w kierunku 5'®3 '. W I. coli, Istnieje pięć polimerazy DNA: polimerazy DNA I i III wykonuje replikację DNA; a polimerazy DNA II, IV i V są odpowiedzialne za replikację i replikację uszkodzonego DNA.
Większość replikacji jest wykonana przez polimerazę DNA III, która jest holoenzymem, który ma 10 różnych podjednostek z kilkoma funkcjami w replikacji DNA. Na przykład podjednostka Alpha jest odpowiedzialna za tworzenie wiązań między nukleotydami.
Kompleks enzymu jest odpowiedzialny za replikację DNA w bakteriach
Helikaza DNA i prima łączy się w celu utworzenia kompleksu zwanego primosoma. To porusza się wzdłuż DNA, działając w skoordynowany sposób, aby oddzielić dwa łańcuchy rodzicielskie, syntetyzując startery w każdym pewnym okresie opóźnionego łańcucha.
Primosom fizycznie wiąże się z polimerazą DNA III i tworzy replisom. Dwie polimerazy DNA III są odpowiedzialne za odtworzenie DNA przewodnika i opóźnionych łańcuchów. W odniesieniu do polimerazy DNA III, opóźniony łańcuch tworzy pętlę, co pozwala dodać nukleotydy do tego łańcucha nastąpiło w tym samym kierunku łańcucha przewodnika.
Dodanie nukleotydów do łańcucha przewodnika jest ciągłe. Podczas opóźnienia jest nieciągłe. Powstają fragmenty 150 nukleotydów o długości, zwane fragmentami Okazaki.
Aktywność egzonukleazy 5 ' -> 3' polimerazy DNA I jest odpowiedzialna za wyeliminowanie starterów i wypełnianie, dodanie nukleotydów. Enzym ligaza uszczelnia szczeliny między fragmentami. Replikacja kończy się, gdy dwie replikacje Hoquillas są w sekwencji ukończenia.
Białko wiąże się z sekwencją zakończenia, zatrzymując ruch widelca replikacji. Topoisomeraza II umożliwia rozdzielenie dwóch chromosomów.
Tryfosforany Desoxiribonukleotydów są stosowane przez polimerazę DNA
Tryfosforan deksynukleozydu (DNTP) zawiera trzy grupy fosforanowe zjednoczone z węglem 5 'deoksyrybosa. DNTPS (DATP, DTTP, DGTP i DCTP) dołączają do łańcucha formy zgodnie z regułą AT/GC.
Może ci służyć: PlesiomorphyPolimeraza DNA katalizuje następującą reakcję: grupa hydroksylowa (-OH) 3 'nukleotydu rosnącego łańcucha reaguje z fosforanem alfa przychodzącego DNTP, uwalniając pirofosforan inorganiczny (PPI) (PPI) (PPI). Hydroliza PPI wytwarza energię do tworzenia wiązania kowalencyjnego lub wiązania fosfodiéster, między nukleotydami rosnącego łańcucha.
Mechanizmy, które zapewniają wierność replikacji DNA
Podczas replikacji DNA polimeraza DNA III popełnia błąd na 100 milionów nukleotydów. Chociaż prawdopodobieństwo błędu jest bardzo niskie, istnieją mechanizmy, które zapewniają wierność w replikacji DNA. Te mechanizmy to:
1) Stabilność w podstawowym apar. Energia wiązania wodorowego między AT/GC jest większa niż w błędnych parach zasad.
2) Struktura aktywnej polimerazy DNA. Polimeraza DNA preferencyjnie katalizuje nukleotydy z prawidłowymi zasadami w przeciwnym łańcuchu. Złe krycie zasad powoduje zniekształcenie podwójnego śmigła DNA, co zapobiega niepoprawnym nukleotydowi zajmującym miejsce aktywnego enzymu.
3) Test czytania. Polimeraza DNA identyfikuje włączone błędne nukleotydy i eliminuje je z łańcucha córki. Aktywność egzonukleazy polimerazy DNA łamie wiązania fosfodiéster między nukleotydami na końcu nowego łańcucha.
Replikacja DNA u eukariotów
W przeciwieństwie do replikacji w prokariotach, których replikacja rozpoczyna się w jednym miejscu, replikacja eukariotyczna zaczyna się w wielu miejscach pochodzenia, a widelca replikacji porusza się dwukierunkowo. Następnie wszystkie widelce replikacji są scalone, tworząc dwa siostrzane chromatydy połączone w centromerze.
Eukarioty mają wiele rodzajów polimerazy DNA, których nazwy używają greckich liter. Polimeraza DNA α tworzyć kompleks z prima. Ten kompleks syntetyzuje krótkie startery składające się z 10 nukleotydów RNA, a następnie od 20 do 30 nukleotydów DNA.
Następnie polimeraza DNA ε albo δ katalizuje wydłużenie łańcucha córki z podkładu. Polimeraza DNA ε Jest zaangażowany w syntezę wiodącego łańcucha, podczas gdy polimeraza DNA δ Syntetyzuj opóźniony łańcuch.
Polimeraza DNA δ Wydłużmy lewy fragment Okazaki, aż dotrze do prawego podkładu RNA, wytwarzając krótkie podnoszenie podkładu („krótka klapa”). W przeciwieństwie do prokariotów, w których polimeraza DNA eliminuje starter, w eukariotach enzym płata endonukleazy eliminuje starter RNA.
Następnie ligaza DNA uszczelnia fragmenty DNA, które są przylegające. Zakończenie replikacji odbywa się wraz z dysocjacją białek widelca replikacji.
Replikacja DNA u eukariotów i cyklu komórkowego
Replikacja u eukariotów występuje w fazie S cyklu komórkowego. Replikowane cząsteczki DNA są segregowane w dwóch komórkach potomnych podczas mitozy. Fazy G1 i G2 oddzielają fazę i mitozę S. Postęp w każdej fazie cyklu komórkowego jest wysoce regulowany przez kinazy, fosfatazy i proteazy.
W fazie G1 cyklu komórkowego kompleks rozpoznawania pochodzenia (OCR) dołącza do miejsca pochodzenia. To indukuje związek helikopoli MCM i innych białek, takich jak CDC6 i CDT1, tworząc kompleks przedreplikacyjny (PRERC). Las helikaza MCM dołącza do łańcucha przewodnika.
Może ci służyć: połączone genyW fazie S PRERR staje się aktywnym miejscem replikacji. Uwalniane są białka OCR, CDC6 i CDT1, a helikaza MCM porusza się w kierunku od 3 'do 5'. Po zakończeniu replikacji zostanie to ponownie uruchomione w następnym cyklu komórkowym.
Replikacja końców chromosomów u eukariotów
Końce chromosomów są znane jako telomery, które składają się z powtarzanych sekwencji w tandemie i regionie 3 ', który wyróżnia się, od 12 do 16 nukleotydów długości.
Polimeraza DNA nie jest w stanie powtórzyć końca łańcuchów DNA 3 '. Wynika to z faktu, że polimeraza DNA może tylko syntetyzować DNA w adresie 5'-3 'i może wydłużyć wcześniej istniejące łańcuchy, bez możliwości syntezy startera w tym regionie. W konsekwencji telomery są skrócone w każdej rundzie replikacji.
Enzym telomerazy zapobiega skracaniu telomerów. Telomeraza jest enzymem, który ma podjednostki białka i RNA (trzeci). Ten ostatni wiąże się z powtarzanymi sekwencjami DNA i pozwala na połączenie telomerazy z końcem 3 '.
Sekwencja RNA za unii zlokalizowanym jako pleśń do syntezy sekwencji sześciu nukleotydów (polimeryzacja) na końcu łańcucha DNA. Wydłużenie telomeru jest katalizowane przez podjednostki telomerazy, zwane odwrotną transkryptazą telomerazy (TERT).
Po polimeryzacji zachodzi transokalizacja, polegająca na ruchu telomerazy do nowego końca łańcucha DNA, łącząc sześć innych nukleotydów do końca.
Funkcje innych polimeraz DNA u eukariontów
Polimeraza DNA β Odgrywa ważną rolę w eliminacji nieprawidłowych zasad DNA, ale nie bierze udziału w replikacji DNA.
Wiele odkrytych polimerazy DNA należy do grupy polimerazy „replikującej translezję”. Te polimerazy są odpowiedzialne za syntezę łańcuchów uzupełniających w uszkodzonym regionie DNA.
Istnieje kilka rodzajów polimeraz „replikacji translezji”. Na przykład polimeraza DNA η Możesz powtórzyć o Timina Dímeros, które są wytwarzane przez UV Light.
Replikacja DNA w archeobakterie
Replikacja DNA Archeobacteria jest podobna do tego podanego w eukariotach. Wynika to z następujących: 1) białka, które uczestniczą w replikacji, są bardziej podobne do białek eukariotów niż do białek prokariotów; oraz 2) Chociaż istnieje tylko jedno miejsce replikacji, jak w prokariotach, jego sekwencja jest podobna do miejsca pochodzenia eukariotycznego.
Podobieństwo w replikacji między łukami i eukariotami potwierdza ideę, że obie grupy są filogenetycznie bardziej ze sobą powiązane niż którekolwiek z nich z prokariotami.
Bibliografia
- Brooker, r. J. 2018. Analiza genetyki i zasady. McGraw-Hill, Nowy Jork.
- Hartwell, L. H., Goldberg, m. L., Fischer, J. DO., Hood, L. 2018. Genetyka - z genów genomów. McGraw-Hill, Nowy Jork.
- Kušić-tišma, j. 2011. Podstawowe aspekty replikacji DNA. Intech Open Access, Chorwacja.
- Lewis, r., 2015. Koncepcje i zastosowania genetyki ludzkiej. McGraw-Hill, Nowy Jork.
- Pierce, ur. DO. 2005. Genetyka - podejście koncepcyjne. W. H. Freeman, Nowy Jork.

