Metody badania Haplootepe, diagnostyka, choroby
- 4529
- 1059
- Pani Gilbert Stolarczyk
A Haplootepe Jest to region genomu, który ma tendencję do dziedziczenia przez wiele pokoleń; Zazwyczaj wszystko znajduje się w tym samym chromosomie. Haplootepy są produktem wiązania genetycznego i pozostają nienaruszone podczas rekombinacji genetycznej.
Słowo „haplotipo” wywodzi się z kombinacji słowa „haploid” i słowa „genotyp”. „Haploid” odnosi się do komórek z jednym zestawem chromosomów, a „genotyp” mówi o składzie genetycznym organizmu.
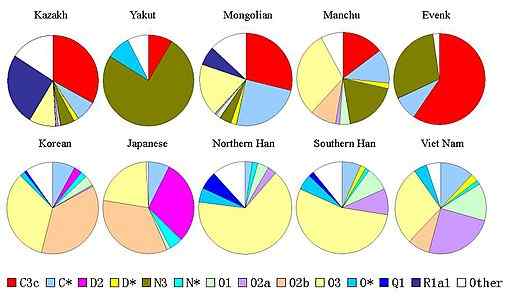
 Schemat rozmieszczenia haplotypów chromosomowych i populacji azjatyckich (źródło: Moogalord [domena publiczna] za pośrednictwem Wikimedia Commons) Po definicji haplotyp może opisać kilka genów lub więcej, które są dziedziczone razem na chromosomie z rodzicielskiego lub może opisać chromosom, który jest całkowicie odziedziczony po rodzicielstwie, podobnie jak w przypadku chromosomu i mężczyzn.
Schemat rozmieszczenia haplotypów chromosomowych i populacji azjatyckich (źródło: Moogalord [domena publiczna] za pośrednictwem Wikimedia Commons) Po definicji haplotyp może opisać kilka genów lub więcej, które są dziedziczone razem na chromosomie z rodzicielskiego lub może opisać chromosom, który jest całkowicie odziedziczony po rodzicielstwie, podobnie jak w przypadku chromosomu i mężczyzn.
Na przykład, gdy haplotypy dzielą geny dla dwóch różnych postaci fenotypowych, takich jak kolor włosów i kolor oczu, osoby posiadające gen koloru włosów będą również posiadać drugi gen dla koloru oczu.
Haplooteypes są jednym z najczęściej używanych narzędzi do badania genealogii, w celu śledzenia pochodzenia chorób, w celu scharakteryzowania zmienności genetycznej i filogeografii populacji różnych rodzajów żywych istot.
Istnieje wiele narzędzi do studiowania halootypes, jeden z najczęściej używanych dzisiaj jest ”Mapa haplotypu„(Hapmap), która jest stroną internetową, która pozwala określić, jakie są segmenty genomu, które są haplotypami.
[TOC]
Metody badań
Haplooteypes stanowią okazję do zrozumienia dziedziczenia genów i ich polimorfizmu. Z odkryciem techniki „reakcja łańcuchowa polimerazy” (PCR, angielski ”Reakcja łańcuchowa polimerazy”) Rozwijał się szeroko w badaniu haplotypów.
Obecnie istnieją wiele metodologii badań haplotypów, niektóre z najważniejszych atrakcji to:
Sekwencjonowanie DNA i wykrywanie polimorfizmu pojedynczego nukleotydu (SNP)
Rozwój technologii sekwencjonowania nowej generacji stanowi wielki skok do badań haplotypów. Nowe technologie umożliwiają różnice do pojedynczej podstawy nukleotydów w określonych regionach haplotypu.
Może ci służyć: rekombinowany DNA: technika, zastosowania i fundamentyW bioinformatyce termin haplotyp jest również używany w odniesieniu do dziedziczenia pojedynczych polimorfizmów nukleotydowych (SNP) w sekwencjach DNA.
Łączenie programów bioinformatycznych z wykrywaniem haplotypów za pomocą sekwencjonowania nowej generacji można dokładnie zidentyfikować pozycję, zastępowanie i wpływ zmiany każdej bazy na genom populacji.
Mikrosatelity (SSR)
Mikrosatelity lub SSR pochodzą z angielskiegowdrożyć powtórzenie I Krótki powtórzenie tandemowe". Są to krótkie sekwencje nukleotydowe, które są powtarzane sukcesywnie w regionie genomu.
Często występuje mikrosatelity wewnątrz haplotypów niekodujących, a zatem poprzez wykrywanie zmian w liczbie powtórzeń mikrosatelitów, różne allele można zaobserwować w haplotypach osobników.
Opracowano markery molekularne mikrosatelitarne do wykrywania niekończących się haplotypów, z płci roślin takich jak papa (Carica Papaya) aż do wykrywania chorób ludzkich, takich jak anemia Falciform.
Polimorfizmy wzmocnionych fragmentów (AFLP)
Ta technika łączy amplifikację z reakcjami PCR z trawieniem DNA z dwoma różnymi enzymami ogranicznymi. Technika wykrywa loci polimorficzne w haplootpy zgodnie z różnymi miejscami cięcia w sekwencji DNA.
Aby lepiej zilustrować tę technikę, wyobraźmy sobie trzy fragmenty tkaniny o tej samej długości, ale pokrojone w różne miejsca (te fragmenty reprezentują trzy fragmenty amplifikowanych haplotypów za pomocą techniki PCR).
W momencie wycięcia materiału, zostanie uzyskanych wielu kawałków różnych rozmiarów, ponieważ każda tkanina jest krojona w różne miejsca. Zamawiając fragmenty zgodnie z rodzajem tkaniny, z którego pochodzą, możemy zaobserwować, gdzie znajdują się różnice między tkaninami lub haplotypami.
Diagnostyka i choroby
Ważną zaletą badań genetycznych haplotypów jest to, że pozostają one prawie nienaruszone lub niezmienione przez tysiące pokoleń, co pozwala na identyfikację odległych przodków i każdej z mutacji, z którymi jednostki przyczyniają się do rozwoju chorób.
Może ci służyć: warianty fenotypoweHaplootepy w ludzkości różnią się w zależności od ras i, zaczynając od tej miarki, geny wykryto w haplotypach, które powodują poważne choroby w każdej z ludzkich ras.
W projekcie Hapmap Uwzględniono cztery grupy rasowe: Europejska, Nigeryjska Joruba, Chińskie Han i Japończycy.
W ten sposób projekt Hapmap Może obejmować różne grupy populacji i śledzić pochodzenie i ewolucję wielu chorób dziedzicznych, które wpływają na każdą z czterech ras.
Jedną z chorób, które najczęściej diagnozuje się za pomocą analizy haplotypów, jest niedokrwistość Falciform u ludzi. Choroba ta jest diagnozowana poprzez śledzenie częstotliwości haplotypów afrykańskich do populacji.
Będąc chorobą pochodzącą z Afryki, identyfikacja afrykańskich haplotypów w populacjach pozwala łatwo śledzić osoby posiadające mutację w sekwencji genetycznej dla globin beta erytrocytów w postaci Hoz (charakterystyczne dla patologii).
Przykłady
Z haplotypami drzew filogenetyczny reprezentujący ewolucyjne zależności między każdym z haplotypów znalezionych w próbie homologicznych cząsteczek DNA lub tym samym gatunkiem, w regionie, który ma niewielką rekombinację lub żaden.
Jedną z najczęściej badanych gałęzi przez haplotypy jest ewolucja układu odpornościowego ludzi. Haplooteypes, który kod kodowania receptora typu Toll (kluczowy element wrodzonego układu odpornościowego) zostały zidentyfikowane dla genomu neandertalskiego i denisovanos.
Pozwala im to prześledzić, jak zmieniły się sekwencje genetyczne w populacjach „współczesnych” ludzi w odniesieniu do sekwencji haplotypowych odpowiadających „przodkom” ludziom.
Budowanie sieci relacji genetycznych z haplotypu mitochondrialnego.
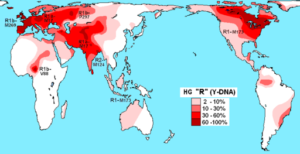 Rozkład haplogrupy r (DNA-Y) w populacjach natywnych (źródło: maucioni [cc przez 3.0 (https: // creativeCommons.Org/licencje/według/3.0)], za pośrednictwem Wikimedia Commons) Różnorodność haplotypów służy do śledzenia i badania różnorodności genetycznej zwierząt podniesionej w niewoli. Techniki te są stosowane specjalnie dla trudnych gatunków do monitorowania dzikiej przyrody.
Rozkład haplogrupy r (DNA-Y) w populacjach natywnych (źródło: maucioni [cc przez 3.0 (https: // creativeCommons.Org/licencje/według/3.0)], za pośrednictwem Wikimedia Commons) Różnorodność haplotypów służy do śledzenia i badania różnorodności genetycznej zwierząt podniesionej w niewoli. Techniki te są stosowane specjalnie dla trudnych gatunków do monitorowania dzikiej przyrody.
Gatunki zwierząt, takie jak rekiny, ptaki i duże ssaki, takie jak jaguary, słonie, są stale genetycznie oceniane za pomocą haplotypów mitochondrialnych w celu monitorowania genetycznego stanu populacji w niewoli niewoli.
Bibliografia
- Bahlo, m., Stankovich, J., Prędkość, t. P., Rubio, J. P., Burfoot, r. K., I stopa, s. J. (2006). Skecowanie wymiany haplotypów genomu za pomocą danych SNP lub mikrosatelitarnego haplotypu. Human Genetics, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., I Kelso, J. (2016). Introgresja haplotypów podobnych do neandertalczyków i Denisovana przyczynia się do adaptacyjnej zmienności w ludzkich odbiornikach podobnych do. The American Journal of Human Genetics, 98 (1), 22-33.
- Z vries, h. G., Van der meulen, m. DO., Rozen, r., Halley, d. J., Scheffer, h., Leo, s.,… I ty Meerman, G. J. (1996). Tożsamość haplotypów między osobami, które dzielą allel mutacji CFTR. Human Genetics, 98 (3), 304-309
- Swallow, m. DO., Leaver, a. L., Christiansen, f. T., Witt, c. S., Abraham, L. J., & Dawkins, r. L. (1992). Haplotypy rodowe: haplotypy populacji Conserva. Ludzka immunologia, 34 (4), 242-252.
- Fellows, m. R., Hartman, t., Hermelin, zm., Landau, g. M., Rosamond, f., I Rozenberg, L. (2009, czerwiec). Wnioskowanie haplotypów ograniczone przez prawdopodobne dane haplotypowe. W corocznym sympozjum na temat kombinatorycznego dopasowywania wzorów (PP. 339-352). Springer, Berlin, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, godz., Moore, J. M., Roy, J., Blumenstiel, ur.,... i liu-cordero, s. N. (2002). Struktura bloków haplotypowych w ludzkim genomie. Science, 296 (5576), 2225-2229.
- Międzynarodowe konsorcjum Hapmap. (2005). Haplotypowa mapa ludzkiego genomu. Nature, 437 (7063), 1299.
- Wynne, r., & Wilding, c. (2018). Mitchondrialne DNA Haplotype Różnorodność i pochodzenie w niewoli piasku Tiger Sharks (Carcharias taurus). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo i. J., Tang, J., Kaslow, r. DO., I Zhang, K. (2007). Wnioskowanie haplotypów dla obecnych danych genotypowych przy użyciu prezydencko zidentyfikowanych haplotypów i wzorców haplotypów. Bioinformatics, 23 (18), 2399-2406.
- Young, n. S. (2018). Anemia aplastyczna. The New England Journal of Medicine, 379 (17), 1643-1656.
- « Charakterystyka dziedziczenia holendrowego, funkcje genów, degeneracja
- Rozczaruj większe cechy, siedlisko, właściwości, opieka »

