Tłumaczenie procesu DNA na eukariot i prokariotów
- 1972
- 377
- Herbert Wróblewski
Tłumaczenie DNA Jest to proces, w którym informacje zawarte w posłańcach wyprodukowane podczas transkrypcji.
Z perspektywy komórkowej ekspresja genu jest stosunkowo złożoną materią, która występuje w dwóch etapach: transkrypcji i translacji.
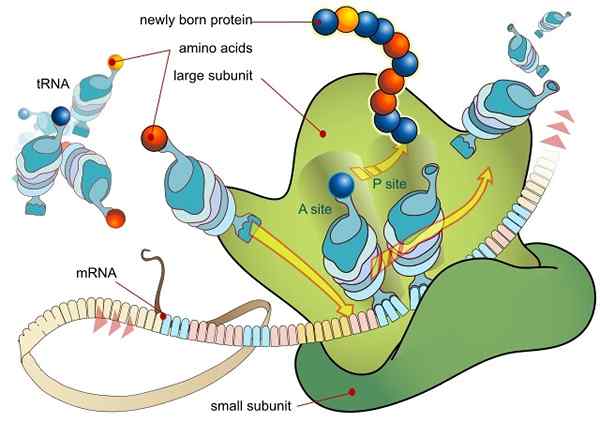
 Tłumaczenie RNA za pośrednictwem rybosomu (Źródło: Ladyofhats / Public Domena, Via Wikimedia Commons)
Tłumaczenie RNA za pośrednictwem rybosomu (Źródło: Ladyofhats / Public Domena, Via Wikimedia Commons) Wszystkie geny, które są wyrażane (niezależnie od tego, czy kodowanie sekwencji peptydowych, to znaczy białka) początkowo robią to poprzez przenoszenie informacji zawartych w jego sekwencji DNA do posłańca Mer Molecula (RNAM) poprzez proces zwany transkrypcją.
Transkrypt osiąga się przez specjalne enzymy znane jako polimerazy RNA, które wykorzystują jedną z komplementarnych nici DNA genu jako pleśni do syntezy cząsteczki „przed arnm”, która następnie jest ścigana w celu utworzenia dojrzałego dojrzałego.
W przypadku genów kodujących białka informacje zawarte w dojrzałym RNM są „odczytane” i tłumaczone w aminokwasach zgodnie z kodem genetycznym, który określa, który potrójka kodonu lub nukleotydu odpowiada każdemu konkretnemu aminokwasowi.
Specyfikacja sekwencji aminokwasowej białka zależy zatem od początkowej sekwencji zasad azotu w DNA, która odpowiada genowi, a następnie w RNA, który transportuje wspomniane informacje z jądra do cytosolu (w komórkach eukariotycznych); proces, który jest również zdefiniowany jako synteza białka prowadzonego przez RNM.
W związku z faktem, że istnieje 64 możliwe kombinacje 4 zasad azotu, które tworzą DNA i RNA oraz tylko 20 aminokwasów, aminokwas może być zakodowany przez różne trzykrotki (kodony), więc mówi się, że kod genetyczny jest ” Zdegeneruj się ”(z wyjątkiem aminokwasowego metioniny, która jest kodowana przez unikalny kodon Aug).
[TOC]
Tłumaczenie eukaryota (stope-procesy)
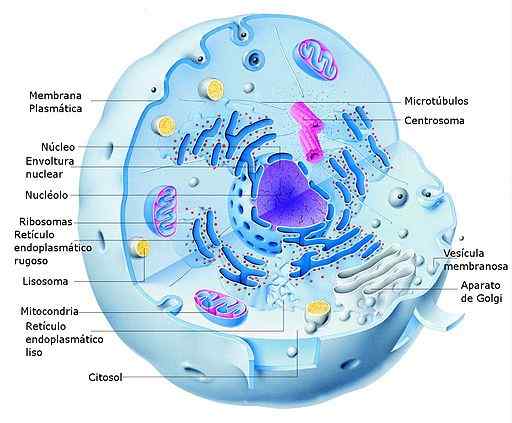
 Schemat komórki eukariotów zwierząt i jej części (źródło: Alejandro Porto [CC0] przez Wikimedia Commons)
Schemat komórki eukariotów zwierząt i jej części (źródło: Alejandro Porto [CC0] przez Wikimedia Commons) W komórkach eukariotycznych transkrypcja ma miejsce w jądrze i tłumaczeniu na cytosol, tak że RNM, które powstają podczas pierwszego procesu, również pełni funkcję w transporcie informacji z jądra do cytosolu, gdzie maszyna biosyntetyczna (rybosomy (rybosomy (rybosomy (rybosomy (rybosomy (rybosomy (rybosomy (rybosomy (rybosomy ).
Ważne jest, aby wspomnieć, że podział transkrypcji i tłumaczenia w eukarionotach jest prawdziwy w przypadku jądra, ale nie jest tak samo w przypadku organelli z własnym genomem, takim jak chloroplasty i mitochondria, które mają bardziej podobne systemy do organizmów prokariotycznych.
Komórki eukariotyczne mają również cytozolowe rybosomy przymocowane do błon retikulum endoplazmatycznego (szorstkie retikulum endoplazmatyczne), w którym występuje translacja białek, które mają być wstawiane w błonach komórkowych lub które wymagają po przetwarzaniu po translacyjnym, które występują we wspomnianym przedziale.
- Przetwarzanie RNM przed tłumaczeniem
RNM są modyfikowane na swoich końcach, gdy są transkrybowane:
- Kiedy 5 'koniec RNM wyłania się z powierzchni polimerazy RNA II podczas transkrypcji, jest to „atakowane” natychmiast przez grupę enzymów, które syntetyzują „maskę” złożoną z 7-metylu guanilado i to jest połączone z terminal nukleotyd RNM poprzez 5 -calowy łącznik, 5 '.
Może ci służyć: kodon- 3 'koniec RNM cierpi z powodu „Clivaje” przez endonukleazę, która generuje wolną grupę hydroksylową 3', która łączy „Ristra” lub „ogon” odpadów adeninowych (od 100 do 250), które są dodawane jednego na tym samym miejscu na tym samym Czas na enzym Poli (A) polimeraza.
„Hood 5” i „ogon” Poli A „Wypełniają funkcje w ochronie cząsteczek RNM przed degradacją, a ponadto działają w transporcie dojrzałych transkryptów do cytosolu oraz w inicjacji i zakończeniu tłumaczenia odpowiednio.
COrte i empalme
Po transkrypcji „pierwotne” RNM z ich dwoma zmodyfikowanymi skrajnościami, wciąż obecnymi w jądrze, przechodzą przez proces „cięcia i splicingu”, w którym sekwencje intronowe są ogólnie eliminowane i powstają powstałe eksony (przetwarzanie po regionalnym), z tym, co uzyskuje się dojrzałe transkrypty, które porzucają jądro i docierają do cytosolu.
Krój i splicing odbywa się przez kompleks ryboproteic o nazwie Espicleosoma (Anglicyzm Spliceosom), utworzone przez pięć małych rybonukleoprotein i cząsteczek RNA, które są zdolne do „rozpoznania” regionów, które należy wyeliminować z pierwotnego transkryptu.
W wielu eukariotach istnieje zjawisko znane jako „cięcie i alternatywne staw”, co oznacza, że różne typy modyfikacji post -rejestracyjnych mogą powodować różne lub izoenzyme, które różnią się od siebie w niektórych aspektach ich sekwencji.
- Rybosomy
Gdy dojrzałe transkrypty opuszczają jądro i są transportowane do translacji na cytosol, są one przetwarzane przez kompleks translacyjny znany jako rybosom, który składa się z kompleksu białkowego związanego z cząsteczkami RNA.

Rybosomy składają się z dwóch podjednostek, jednego „dużego”, a drugiego „małego”, które są swobodnie zdysocjowane w cytosolu i łączą lub kojarzą się na cząsteczce mRNA, która tłumaczy się.
Związek między rybosomami a mRNA zależy od wyspecjalizowanych cząsteczek RNA, które są związane z białkami rybosomalnymi (rybosomalny RNA lub RNA i transfer lub arnt RNA), z których każde wykonuje określone funkcje.
ARNT są molekularnymi „adapterami”, ponieważ przez jeden ze swoich celów mogą „odczytać” każdy kodon lub triplet w dojrzałym RNA (przez komplementarność zasad), a przez drugie mogą dołączyć do aminokwasu zakodowanego przez kodon „czytaj”.
Z drugiej strony cząsteczki RNR są odpowiedzialne za przyspieszanie (katalizowanie) proces wiązania każdego aminokwasu w powstającym łańcuchu peptydowym.
Eukariotyczne dojrzałe RNM można „odczytać” przez wiele rybosomów, tyle razy, ile wskazuje komórka. Innymi słowy, ten sam RNM może prowadzić do wielu kopii tego samego białka.
Rozpoczęcie kodonu i ramy odczytu
Gdy podjednostki rybosomalne podjednostki podjednostka do dojrzałego RNM, kompleks ryboproteic „skanuje” sekwencję wspomnianej cząsteczki, dopóki nie znajdzie kodonu początkowego, co jest zawsze Aug i implikuje wprowadzenie pozostałości metioniny.
Może ci służyć: monoploidia: jak to się dzieje, organizmy, częstotliwość i użytecznośćKodon AUR definiuje ramkę odczytu dla każdego genu, a ponadto definiuje pierwszy aminokwas wszystkich białek przetłumaczonych na naturę (ten aminokwas jest często eliminowany po translacyjnie).
Kodony terminowe
Trzy inne kodony zostały zidentyfikowane jako te, które wywołują zakończenie tłumaczeni: UAA, UAG i UGA.
Te mutacje, które sugerują zmianę zasad azotu w tryplecie, która koduje aminokwas i powodują, że w kodonach zakończenia są znane jako mutacje bezsensowne, ponieważ powodują przedwczesne zatrzymanie procesu syntezy, który tworzy krótsze krótsze białka.
Regiony nietranslacja
W pobliżu końca 5 'dojrzałych cząsteczek RNM istnieją regiony, które nie są przetłumaczone (UTR, z angielskiego Region nietranslatujący), zwane także sekwencjami „lidera”, które znajdują się między pierwszym nukleotydem a początkiem tłumaczenia (sierpień).
Te regiony UTR, które nie są przetłumaczone, mają określone miejsca dla zjednoczenia z rybosomami i ludźmi, na przykład mają przybliżoną długość 170 nukleotydów, w tym regiony regulacyjne, miejsca wiązania białka, które działają w regulacji translacji itp.
- Początek tłumaczenia
Tłumaczenie, a także transkrypcja składa się z 3 faz: jedna z inicjacji, kolejna wydłużenie i wreszcie zakończenie.
Inicjacja
Składa się z montażu kompleksu translacyjnego na RNM, który zasługuje na połączenie trzech białek znanych jako czynniki inicjacyjne (jeśli angielskie Współczynnik inicjacji) IF1, IF2 i IF3 do małej podjednostki rybosomu.
Kompleks „wstępnego inicjacji” utworzony przez czynniki inicjacji i z kolei małe podjednostki rybosomalne, z arntem, że „ładuje” resztę metaoninową, a ten zestaw cząsteczek wiąże.
Wydarzenia te prowadzą do Unii RNM z dużą podjednostką rybosomalną, co prowadzi do uwolnienia czynników inicjacyjnych. Duża podjednostka rybosomu ma 3 miejsca unii dla cząsteczek ARNT: miejsce A (aminokwas), miejsce P (polipeptyd) i miejsce E (wyjście).
Witryna A dołącza do antykodu aminoacilu, który jest uzupełniający się do tłumaczonego mRNA; Miejsce P to miejsce, w którym aminokwas jest przenoszony z ARNT do powstającego peptydu, a miejsce S znajduje.
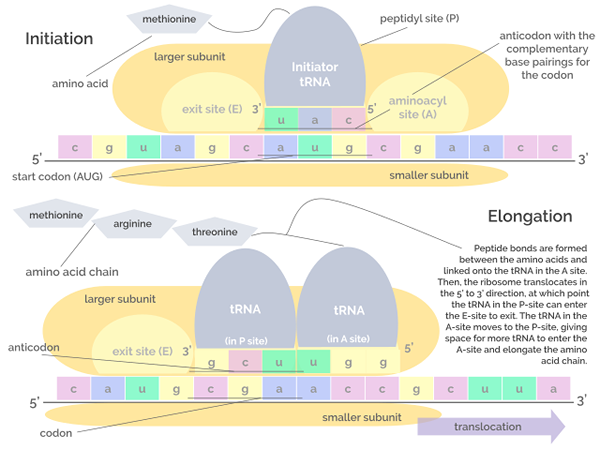 Graficzna reprezentacja faz inicjacji i wydłużenia tłumaczenia (źródło: Jordan Nguyen/CC BY-S (https: // creativeCommons.Org/licencje/nabrzeże/4.0) Via Wikimedia Commons)
Graficzna reprezentacja faz inicjacji i wydłużenia tłumaczenia (źródło: Jordan Nguyen/CC BY-S (https: // creativeCommons.Org/licencje/nabrzeże/4.0) Via Wikimedia Commons) Wydłużenie
Faza ta składa się z „ruchu” rybosomu wzdłuż cząsteczki mRNA i tłumaczenia każdego kodonu, które „czytanie” to, co implikuje wzrost lub wydłużenie łańcucha polipeptydowego podczas urodzenia.
Proces ten wymaga czynnika znanego jako współczynnik wydłużenia GTP w postaci GTP, który jest tym, który napędza translokację współczynników wydłużania wzdłuż cząsteczki RNM podczas tłumaczenia.
Może ci służyć: fragmenty okazakiAktywność peptydylu transferazy rybosomalnych RNA pozwala na tworzenie wiązań peptydowych między kolejnymi aminokwasami, które są dodawane do łańcucha.
Zakończenie
Tłumaczenie kończy się, gdy rybosom spełnia którykolwiek z kodonów zakończenia, ponieważ ARN nie rozpoznają tych kodonów (nie kodują aminokwasów). Połączone są również białka znane jako czynniki uwalniania, które ułatwiają oderwanie rybosoma MR.
Tłumaczenie prokuratora (procesy podania)
W prokariotach, podobnie jak w komórkach eukariotycznych, rybosomy odpowiedzialne za syntezę białka występują w cytosolu (co jest również prawdziwe w przypadku maszyn transkrypcyjnych), co pozwala na szybki wzrost stężenia cytozolowego białka, gdy wyrażanie genów, które tego kodyfikuje to wzrasta.
Chociaż nie jest to niezwykle powszechny proces w tych organizmach, pierwotne RNM wytwarzane podczas transkrypcji może ponieść dojrzewanie po regulacji poprzez „cięcie i splicing”. Jednak najczęstszym obserwowaniem rybosomów przymocowanych do pierwotnego transkrybowanego, które tłumaczą je w tym samym czasie, gdy jest to transkrybowane z odpowiedniej sekwencji DNA.
W związku z powyższym tłumaczenie w wielu prokariotach zaczyna się od końca 5 ', ponieważ koniec 3' mRNA pozostaje powiązany z DNA formy (i występuje jednocześnie z transkrypcją).
Regiony nietranslacja
Komórki prokariotyczne wytwarzają również RNM z nietranslacjami, które są znane jako „Shine-Dalgarno Box” i którego sekwencja konsensusowa jest AGGAGG. Jak widać, regiony UTR bakterii są znacznie krótsze niż w komórkach eukariotycznych, chociaż wykonują podobne funkcje podczas translacji.
Proces
W bakteriach i innych organizmach prokariotycznych proces translacji jest dość podobny do procesu komórek eukariotycznych. Składa się również z trzech faz: inicjacji, wydłużenia i zakończenia, które zależą od określonych czynników prokariotycznych, różniących się od tych stosowanych przez eukariotów.
Na przykład wydłużenie zależy od czynników wydłużenia zwanych EF-TU i EF-TS, zamiast czynnika eukariotycznego G.
Bibliografia
- Alberts, ur., Johnson, a., Lewis, J., Raff, m., Roberts, k., & Walter, p. (2007). Biologia komórki molekularnej. Garland Science. Nowy Jork, 1392.
- Clay, s. & Brown, w. (2008) Tłumaczenie: DNA na mRNA na białko. Nature Education 1 (1): 101.
- Griffiths, a. J., Wessler, s. R., Lewontin, r. C., Gelbart, w. M., Suzuki, zm. T., & Miller, j. H. (2005). Wprowadzenie do analizy genetycznej. Macmillan.
- Lodish, h., Berk, a., Kaiser, c. DO., Krieger, m., Scott, m. P., Bretscher, a.,… & Matsudaira, p. (2008). Biologia komórek molekularnych. Macmillan.
- Nelson, zm. L., Lehninger, a. L., & Cox, m. M. (2008). Zasady biochemii lehninger. Macmillan.
- Rosenberg, L. I., & Rosenberg, D. D. (2012). Ludzkie geny i genomy: nauka. Zdrowie, społeczeństwo, 317-338.
- « Charakterystyka sorbus aria, siedlisko, właściwości, kultura
- Boletus Aereus Cechy, siedlisko, identyfikacja, przepisy »

